"Il recente sviluppo delle tecnologie di sequenziamento ad alta processività (HTS), chiamato anche sequenziamento di nuova generazione (NGS) o sequenziamento "profondo" (deep sequencing), che ha rivoluzionato le conoscenze sulla genomica degli organismi animali e vegetali, sta penetrando sempre più il campo della diagnostica fitopatologica, con un'enfasi particolare per i virus vegetali, ormai studiati da quasi un decennio". Questa la sintesi di una recente pubblicazione (luglio 2018) apparsa sul Bollettino dell'EPPO (Organizzazione Europea per la Protezione delle Piante), che preannuncia l'inizio di una nuova era nella diagnosi di batteri,funghi,virus e viroidi delle piante.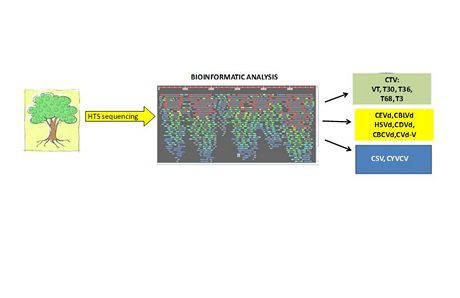 Figura 1 (qui sopra).Schema sintetico delle diverse fasi di sequenziamento, allineamento dei frammenti di sequenza e confronto con genomi in banca dati.
Figura 1 (qui sopra).Schema sintetico delle diverse fasi di sequenziamento, allineamento dei frammenti di sequenza e confronto con genomi in banca dati.
"L'impiego di queste tecnologie - spiegano, tra l'altro gli autori, darà un contributo importante alla protezione delle colture e alla prevenzione e gestione delle emergenze fitosanitarie".
A commentare la notizia è Grazia Licciardello, biologo molecolare presso l'AgroBioTech di Catania, (laboratorio accreditato dal Servizio Fitosanitario Regionale per la diagnosi fitopatologica).
"I controlli sanitari delle piante non verrebbero più effettuate attraverso singole analisi, specifiche per ogni patogeno - spiega la biologa - ma attraverso il sequenziamento del genoma di tutti gli organismi presenti, generando una massa enorme di dati, che sono poi opportunamente elaborati mediante analisi bioinformatica".
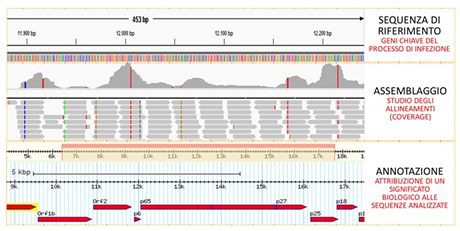 Grafico sopra: fasi della ricostruzione del genoma del virus della tristezza degli agrumi. Il processo si avvale di banche dati che contengono le sequenze nucleotidiche di altri genomi del virus già noti. L'assemblaggio avviene allineando le frazioni di sequenze ottenute rispetto al/i genoma/i di riferimento e verificando il livello di copertura. Lo studio delle sequenze analizzate (annotazione) permette poi di individuare i singoli geni. (Clicca qui per ingrandimento)
Grafico sopra: fasi della ricostruzione del genoma del virus della tristezza degli agrumi. Il processo si avvale di banche dati che contengono le sequenze nucleotidiche di altri genomi del virus già noti. L'assemblaggio avviene allineando le frazioni di sequenze ottenute rispetto al/i genoma/i di riferimento e verificando il livello di copertura. Lo studio delle sequenze analizzate (annotazione) permette poi di individuare i singoli geni. (Clicca qui per ingrandimento)
"Nel caso di virus e viroidi - continua Licciardelli - si procede all'estrazione dell'RNA da porzioni di corteccia, al sequenziamento NGS e all'analisi bioinformatica. Le sequenze ottenute sono assemblate come i pezzi di un puzzle per ricostruire l'intero genoma di tutti i virus e viroidi presenti, attraverso un'analisi computazionale di multi-allineamento con i genomi presenti nelle banche dati. In tal modo è possibile non solo distinguere la specie ma anche gli isolati e i ceppi dei patogeni , se già noti, e di rilevare la presenza di organismi imprevisti o sconosciuti che potrebbero costituire potenziali minacce".
"Questo tipo di analisi 'metagenomica' si sta diffondendo rapidamente - aggiunge dal canto suo Antonino Catara, già presidente dell' Organizzazione Internazionale dei Virologi degli Agrumi e ordinario di Patologia vegetale all'Università di Catania – in quanto consente il rilevamento di qualsiasi organismo presente in un campione. La sua applicazione offre nuove possibilità diagnostiche nei programmi di sorveglianza, nella certificazione del materiale di propagazione vegetale, nei controlli di quarantena per evitare l'introduzione di parassiti esotici e, infine, nel monitoraggio del materiale importato".
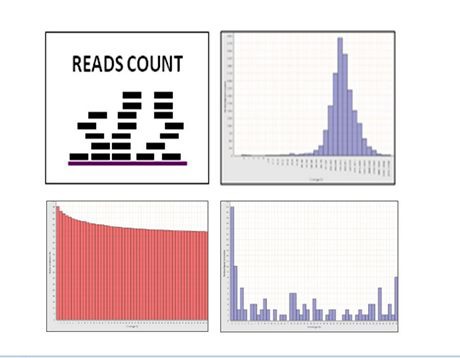 Figura 2.Rappresentazione grafica delle fasi di verifica bioinformatica: conteggio dei frammenti di sequenza e analisi qualitative del livello di sovrapposizione con genomi di riferimento.
Figura 2.Rappresentazione grafica delle fasi di verifica bioinformatica: conteggio dei frammenti di sequenza e analisi qualitative del livello di sovrapposizione con genomi di riferimento.
Dopo una lunga fase di messa a punto, iniziata dal Parco Scientifico e Tecnologico della Sicilia nell'ambito del progetto PON IT-Citrus genomics, la tecnologia è stata testata da AgroBioTech nella certificazione 'virus esentè su materiale di propagazione degli agrumi, confermando di poter di rilevare 'simultaneamentè (per confronto) tutti i virus e viroidi noti presenti nelle banche dati.
"Secondo le Organizzazioni Internazionali questa piccola ma grande rivoluzione - conclude il cattedratico - comporterà vantaggi notevoli per i Servizi Fitosanitari, i vivaisti e i produttori, faciliterà la circolazione di nuove varietà (perché "controllate ai raggi X"), e semplificherà, razionalizzandoli,i numerosi monitoraggi oggi previsti. Effetti positivi sono attesi anche sull'occupazione di nuove professionalità, da affiancare ad agronomi, biologi e biotecnologi, potenziando l'efficienza del sistema. Va da sé che occorrerebbe predisporre strumenti, strutture e competenze adeguate".
